8.1 Análise de Componentes Principais (PCA)
A Análise de Componentes Principais (PCA na sigla em inglês) é uma técnica de redução de dimensionalidade usualmente aplicada a um grande número de variáveis relacionadas, de forma a capturar o máximo possível da variabilidade do conjunto de dados. Foi introduzida por (Pearson 1901) e estudada independentemente por (Hotelling 1933) e outros pesquisadores que abordaram o problema de formas variadas. Considerando a definição de (Bishop 1999), seja um conjunto de dados \(X\) de dimensão \(n \times p\) composto por \(n\) vetores \(p\)-dimensionais conforme indicado a seguir. \[ X = \begin{bmatrix} x_{1,1} & x_{1,2} & \cdots & x_{1,p} \\ x_{2,1} & x_{2,2} & \cdots & x_{2,p} \\ \vdots & \vdots & \ddots & \vdots \\ x_{n,1} & x_{n,2} & \cdots & x_{n,p} \end{bmatrix} = \begin{bmatrix} \boldsymbol{x}_{1} \\ \boldsymbol{x}_{2} \\ \vdots \\ \boldsymbol{x}_{n} \end{bmatrix} \]
8.1.1 Via matriz de covariâncias \(S\)
Deste conjunto de dados calcula-se a matriz de covariâncias amostrais \(S\) conforme Eq. (4.17). São obtidos os autovalores \(\lambda\) associados aos autovetores \(\boldsymbol{v}_i \ne \boldsymbol{0}\) da matriz \(S\) pela equação (3.15), \(i=1,\ldots,p\). Os autovetores correspondentes aos \(q\) maiores autovetores (\(q<p\)) são mantidos, e uma representação de dimensão reduzida é definida por uma combinação linear dos autovetores e dos dados deslocados pela média.
Exemplo 8.1 Pode-se calcular os autovetores e autovalores das colunas numéricas do banco de dados iris.
df <- iris # simplificando/padronizando o nome da base de dados
(m <- colMeans(df[-5])) # vetor de medias## Sepal.Length Sepal.Width Petal.Length Petal.Width
## 5.843333 3.057333 3.758000 1.199333## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 0.6856935 -0.0424340 1.2743154 0.5162707
## Sepal.Width -0.0424340 0.1899794 -0.3296564 -0.1216394
## Petal.Length 1.2743154 -0.3296564 3.1162779 1.2956094
## Petal.Width 0.5162707 -0.1216394 1.2956094 0.5810063## eigen() decomposition
## $values
## [1] 4.22824171 0.24267075 0.07820950 0.02383509
##
## $vectors
## [,1] [,2] [,3] [,4]
## [1,] 0.36138659 0.65658877 0.58202985 0.3154872
## [2,] -0.08452251 0.73016143 -0.59791083 -0.3197231
## [3,] 0.85667061 -0.17337266 -0.07623608 -0.4798390
## [4,] 0.35828920 -0.07548102 -0.54583143 0.7536574## Standard deviations (1, .., p=4):
## [1] 2.0562689 0.4926162 0.2796596 0.1543862
##
## Rotation (n x k) = (4 x 4):
## PC1 PC2 PC3 PC4
## Sepal.Length 0.36138659 -0.65658877 0.58202985 0.3154872
## Sepal.Width -0.08452251 -0.73016143 -0.59791083 -0.3197231
## Petal.Length 0.85667061 0.17337266 -0.07623608 -0.4798390
## Petal.Width 0.35828920 0.07548102 -0.54583143 0.7536574Pode-se investigar o objeto av.
## [1] "sdev" "rotation" "center" "scale" "x"## [1] "prcomp"## PC1 PC2 PC3 PC4
## Sepal.Length 0.36138659 -0.65658877 0.58202985 0.3154872
## Sepal.Width -0.08452251 -0.73016143 -0.59791083 -0.3197231
## Petal.Length 0.85667061 0.17337266 -0.07623608 -0.4798390
## Petal.Width 0.35828920 0.07548102 -0.54583143 0.7536574Mais alguns exercícios.
# Calculando a primeira componente principal 'na mão'
pc1 <- av$rotation[1,1]*df[,1] +
av$rotation[2,1]*df[,2] +
av$rotation[3,1]*df[,3] +
av$rotation[4,1]*df[,4]
# colando e removendo pc1
df <- cbind(iris, pc1)
rm(pc1)
# dando uma olhada
head(df)## Sepal.Length Sepal.Width Petal.Length Petal.Width Species pc1
## 1 5.1 3.5 1.4 0.2 setosa 2.818240
## 2 4.9 3.0 1.4 0.2 setosa 2.788223
## 3 4.7 3.2 1.3 0.2 setosa 2.613375
## 4 4.6 3.1 1.5 0.2 setosa 2.757022
## 5 5.0 3.6 1.4 0.2 setosa 2.773649
## 6 5.4 3.9 1.7 0.4 setosa 3.221505
## [1] TRUE## [1] TRUE8.1.2 Via matriz de correlação \(R\)
É possível realizar o mesmo procedimento na matriz de correlação \(R\) dada pela Eq. (4.24). Esta abordagem é recomendada para evitar que os resultados sejam afetados pela escala dos valores observados.
## Sepal.Length Sepal.Width Petal.Length Petal.Width pc1
## Sepal.Length 1.0000000 -0.1175698 0.8717538 0.8179411 0.8974018
## Sepal.Width -0.1175698 1.0000000 -0.4284401 -0.3661259 -0.3987485
## Petal.Length 0.8717538 -0.4284401 1.0000000 0.9628654 0.9978739
## Petal.Width 0.8179411 -0.3661259 0.9628654 1.0000000 0.9665475
## pc1 0.8974018 -0.3987485 0.9978739 0.9665475 1.0000000## eigen() decomposition
## $values
## [1] 3.911658e+00 9.166484e-01 1.474599e-01 2.423405e-02 7.018110e-16
##
## $vectors
## [,1] [,2] [,3] [,4] [,5]
## [1,] 0.4522259 -0.356806670 0.73993342 -0.3275283 -0.11577781
## [2,] -0.2227481 -0.932011105 -0.24995244 0.1380551 0.01425325
## [3,] 0.5026424 -0.003345711 -0.12406502 0.6241962 -0.58508668
## [4,] 0.4887010 -0.045988343 -0.60912426 -0.6138891 -0.10566043
## [5,] 0.5043778 -0.043797032 -0.05998183 0.3273916 0.79555125## Standard deviations (1, .., p=5):
## [1] 1.977791e+00 9.574176e-01 3.840051e-01 1.556729e-01 4.922447e-16
##
## Rotation (n x k) = (5 x 5):
## PC1 PC2 PC3 PC4 PC5
## Sepal.Length 0.4522259 -0.356806670 0.73993342 -0.3275283 0.11577781
## Sepal.Width -0.2227481 -0.932011105 -0.24995244 0.1380551 -0.01425325
## Petal.Length 0.5026424 -0.003345711 -0.12406502 0.6241962 0.58508668
## Petal.Width 0.4887010 -0.045988343 -0.60912426 -0.6138891 0.10566043
## pc1 0.5043778 -0.043797032 -0.05998183 0.3273916 -0.795551258.1.3 Proporção da variância explicada
A proporção da variância explicada pelo \(i\)-ésimo componente principal é dada pela Eq. (8.1), e pode ser visualizada em um gráfico ordenado, o screeplot proposto por (Cattell 1966) para testar o número de fatores. \[\begin{equation} PVE_i = \dfrac{\lambda_i}{\sum_{j=1}^{p} \lambda_j} \tag{8.1} \end{equation}\]
## [1] 4.22824171 0.24267075 0.07820950 0.02383509## [1] 0.924618723 0.053066483 0.017102610 0.005212184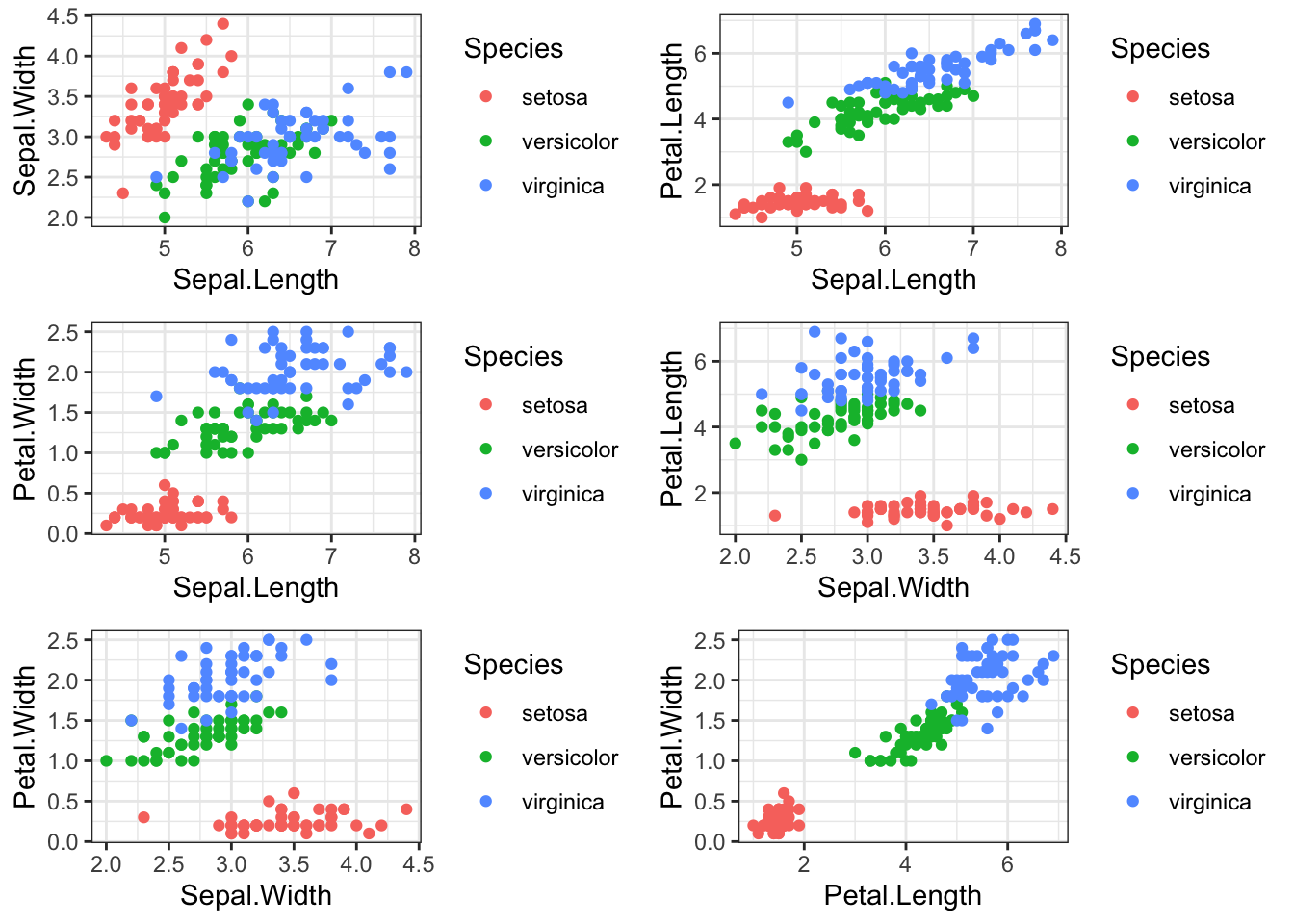
## [1] 3.911658e+00 9.166484e-01 1.474599e-01 2.423405e-02 7.018110e-16## [1] 7.823315e-01 1.833297e-01 2.949199e-02 4.846810e-03 1.403622e-16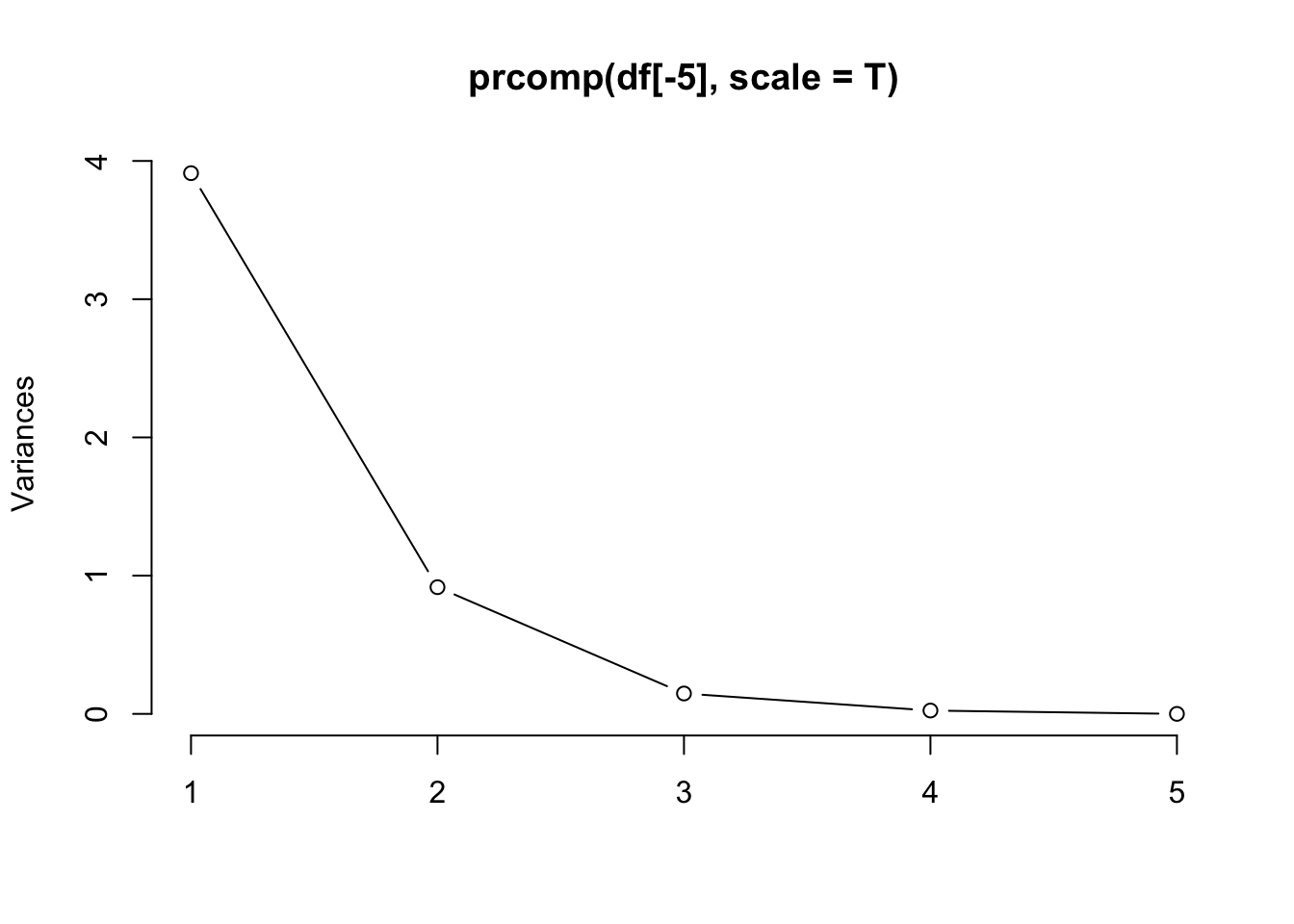
Exemplo 8.2 Considere o banco de dados iris, que contém 4 colunas numéricas com as larguras e comprimentos das pétalas e sépalas de três espécies de lírio (iris).
## Sepal.Length Sepal.Width Petal.Length Petal.Width Species
## 1 5.1 3.5 1.4 0.2 setosa
## 2 4.9 3.0 1.4 0.2 setosa
## 3 4.7 3.2 1.3 0.2 setosa
## 4 4.6 3.1 1.5 0.2 setosa
## 5 5.0 3.6 1.4 0.2 setosa
## 6 5.4 3.9 1.7 0.4 setosaExistem \({4 \choose 2} = 6\) combinações possíveis de gráficos bidimensionais, apresentados a seguir.
require(gridExtra)
p1 <- ggplot(iris, aes(Sepal.Length, Sepal.Width, colour = Species)) + geom_point()
p2 <- ggplot(iris, aes(Sepal.Length, Petal.Length, colour = Species)) + geom_point()
p3 <- ggplot(iris, aes(Sepal.Length, Petal.Width, colour = Species)) + geom_point()
p4 <- ggplot(iris, aes(Sepal.Width, Petal.Length, colour = Species)) + geom_point()
p5 <- ggplot(iris, aes(Sepal.Width, Petal.Width, colour = Species)) + geom_point()
p6 <- ggplot(iris, aes(Petal.Length, Petal.Width, colour = Species)) + geom_point()
grid.arrange(p1, p2, p3, p4, p5, p6, ncol = 2)
É posível utilizar o método de componentes principais para aprimorar a visualização8 da estrutura de associação entre as diferentes espécies de plantas.
library(ggfortify)
autoplot(prcomp(df[-5]), data = iris, colour = 'Species', shape = 'Species',
loadings = T, loadings.label = T, loadings.label.vjust = 2, type = 'raw')
autoplot(prcomp(df[-5], scale = T), data = iris, colour = 'Species', shape = 'Species',
loadings = T, loadings.label = T)
Exercício 8.1 Considere o banco de dados sobre câncer de mama apresentado por (Dua and Graff 2019). A partir do código abaixo, faça uma anáise de componentes principais desconsiderando as duas primeiras colunas, que indicam respectivamente o código de identificação da paciente (V1) e o diagnóstico (V2, Benigno/Maligno).
(a) Quais os valores de \(n\) e \(p\)?
(b) O que ocorre no comando x2 <- x[,-c(1,2)]?
(c) Obtenha os autovalores e autovetores.
(d) Apresente o screeplot.
(e) Apresente o gráfico dos dois primeiros componentes principais colorido por V2.
(f) Você considera que é possível associar os diagnósticos às variáveis V3 a V32? Dica: observe se há algum tipo de agrupamento no gráfico do item (e).
(g) Quais variáveis mais influenciam nos compomentes principais 1 e 2? Dica: use loadings.label = T na função autoplot do item (e) e observe o gráfico.
library(ggfortify)
x <- read.table('https://filipezabala.com/data/wdbc.data', sep = ',')
x2 <- x[,-c(1,2)]Exercício 8.2 Faça a análise de componentes principais considerando o banco de dados apresentado por (Fokoue 2020). Considere o código a seguir.
tf <- paste0(tempfile(), '.zip')
url0 <- 'https://archive.ics.uci.edu/static/public/518/speaker+accent+recognition.zip'
download.file(url0, tf)
unzip(tf, list = TRUE)
dt <- read.csv(unzip(tf, 'accent-mfcc-data-1.csv'))
summary(dt)
str(dt)
pc <- prcomp(dt[-1])
library(ggfortify)
autoplot(pc, data = dt, colour = 'language',
loadings = T, loadings.label = T, type = 'raw')Exercício 8.3 Faça a análise de componentes principais considerando o banco de dados robCompositions::arcticLake.
References
Exemplos baseados em https://cran.r-project.org/web/packages/ggfortify/vignettes/plot_pca.html.↩︎