9.10 Análise Discriminante
Análise discriminante ou discriminação tem suas origens nos trabalhos de Ronald Fisher (R. A. Fisher 1936), (R. A. Fisher 1938), (R. A. Fisher 1940), e refere-se a um conjunto técnicas multivariadas com o objetivo de separar grupos de observações. É baseada em métodos multivariados, considerando um conjunto de equações de predição baseadas em variáveis independentes. Há dois enfoques típicos em uma análise discriminante:
Classificação, com o intuito de encontrar uma equação preditiva para atribuir novos indivíduos a grupos pré estabelecidos
Discriminação, que tem por objetivo interpretar uma equação preditiva para entender melhor as relações entre as variáveis
A análise discriminante é similar a uma Regressão Linear Múltipla, sendo a que a principal diferença entre estas duas técnicas é que a análise de regressão lida com uma variável dependente contínua, enquanto a análise discriminante deve ter uma variável dependente categórica ou discreta.
9.10.1 Análise Discriminante Linear (LDA)
A LDA assume que os preditores possuem distribuição gaussiana e que as diferentes classes têm médias específicas de classe e mesma (matriz de) variância e covariâncias.
Exemplo 9.20 Classificando espécies de lírio via LDA.
Avaliando a suposição de normalidade por grupo através do teste de Shapiro-Wilk.
## iris[, 5]: setosa
##
## Shapiro-Wilk normality test
##
## data: dd[x, ]
## W = 0.9777, p-value = 0.4595
##
## ---------------------------------------------------------------------------------
## iris[, 5]: versicolor
##
## Shapiro-Wilk normality test
##
## data: dd[x, ]
## W = 0.97784, p-value = 0.4647
##
## ---------------------------------------------------------------------------------
## iris[, 5]: virginica
##
## Shapiro-Wilk normality test
##
## data: dd[x, ]
## W = 0.97118, p-value = 0.2583## iris[, 5]: setosa
##
## Shapiro-Wilk normality test
##
## data: dd[x, ]
## W = 0.97172, p-value = 0.2715
##
## ---------------------------------------------------------------------------------
## iris[, 5]: versicolor
##
## Shapiro-Wilk normality test
##
## data: dd[x, ]
## W = 0.97413, p-value = 0.338
##
## ---------------------------------------------------------------------------------
## iris[, 5]: virginica
##
## Shapiro-Wilk normality test
##
## data: dd[x, ]
## W = 0.96739, p-value = 0.1809## iris[, 5]: setosa
##
## Shapiro-Wilk normality test
##
## data: dd[x, ]
## W = 0.95498, p-value = 0.05481
##
## ---------------------------------------------------------------------------------
## iris[, 5]: versicolor
##
## Shapiro-Wilk normality test
##
## data: dd[x, ]
## W = 0.966, p-value = 0.1585
##
## ---------------------------------------------------------------------------------
## iris[, 5]: virginica
##
## Shapiro-Wilk normality test
##
## data: dd[x, ]
## W = 0.96219, p-value = 0.1098## iris[, 5]: setosa
##
## Shapiro-Wilk normality test
##
## data: dd[x, ]
## W = 0.79976, p-value = 8.659e-07
##
## ---------------------------------------------------------------------------------
## iris[, 5]: versicolor
##
## Shapiro-Wilk normality test
##
## data: dd[x, ]
## W = 0.94763, p-value = 0.02728
##
## ---------------------------------------------------------------------------------
## iris[, 5]: virginica
##
## Shapiro-Wilk normality test
##
## data: dd[x, ]
## W = 0.95977, p-value = 0.08695Avaliando a suposição da igualdade da matriz de variâncias e covariâncias através do teste M de Box (G. E. Box 1949), (G. E. Box 1950). A hipótese sendo testada é \(H_0: \boldsymbol{\Sigma}_1=\boldsymbol{\Sigma}_2=\cdots=\boldsymbol{\Sigma}_k\).
## iris[, 5]: setosa
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 0.12424898 0.099216327 0.016355102 0.010330612
## Sepal.Width 0.09921633 0.143689796 0.011697959 0.009297959
## Petal.Length 0.01635510 0.011697959 0.030159184 0.006069388
## Petal.Width 0.01033061 0.009297959 0.006069388 0.011106122
## ---------------------------------------------------------------------------------
## iris[, 5]: versicolor
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 0.26643265 0.08518367 0.18289796 0.05577959
## Sepal.Width 0.08518367 0.09846939 0.08265306 0.04120408
## Petal.Length 0.18289796 0.08265306 0.22081633 0.07310204
## Petal.Width 0.05577959 0.04120408 0.07310204 0.03910612
## ---------------------------------------------------------------------------------
## iris[, 5]: virginica
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 0.40434286 0.09376327 0.30328980 0.04909388
## Sepal.Width 0.09376327 0.10400408 0.07137959 0.04762857
## Petal.Length 0.30328980 0.07137959 0.30458776 0.04882449
## Petal.Width 0.04909388 0.04762857 0.04882449 0.07543265## iris[, 5]: setosa
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 1.0000000 0.7425467 0.2671758 0.2780984
## Sepal.Width 0.7425467 1.0000000 0.1777000 0.2327520
## Petal.Length 0.2671758 0.1777000 1.0000000 0.3316300
## Petal.Width 0.2780984 0.2327520 0.3316300 1.0000000
## ---------------------------------------------------------------------------------
## iris[, 5]: versicolor
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 1.0000000 0.5259107 0.7540490 0.5464611
## Sepal.Width 0.5259107 1.0000000 0.5605221 0.6639987
## Petal.Length 0.7540490 0.5605221 1.0000000 0.7866681
## Petal.Width 0.5464611 0.6639987 0.7866681 1.0000000
## ---------------------------------------------------------------------------------
## iris[, 5]: virginica
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## Sepal.Length 1.0000000 0.4572278 0.8642247 0.2811077
## Sepal.Width 0.4572278 1.0000000 0.4010446 0.5377280
## Petal.Length 0.8642247 0.4010446 1.0000000 0.3221082
## Petal.Width 0.2811077 0.5377280 0.3221082 1.0000000##
## Box's M-test for Homogeneity of Covariance Matrices
##
## data: iris[, -5]
## Chi-Sq (approx.) = 140.94, df = 20, p-value < 2.2e-16A diferença significativa (p-value < 2.2e-16) sugere que é mais interessante considerar a QDA, que não faz a suposição de igualdade de matrizes de variâncias e covariâncias. De toda forma procederemos com a LDA para comparação. Inicialmente é obtida a amostra de treino. O número máximo de funções discriminantes úteis que podem separar os \(G\) grupos é o mínimo entre \(G-1\) e \(p\), logo \(\text{min}(3-1,4)=2\).
# amostra de treino: 60%
set.seed(1); itrain <- sort(sample(1:nrow(iris), floor(.6*nrow(iris))))
train <- iris[itrain,]
test <- iris[-itrain,]
table(train$Species)##
## setosa versicolor virginica
## 32 26 32O ajuste do modelo de análise discriminante linear pode ser feito pela função MASS::lda.
## Call:
## lda(Species ~ ., data = iris, prior = c(1, 1, 1)/3, subset = itrain)
##
## Prior probabilities of groups:
## setosa versicolor virginica
## 0.3333333 0.3333333 0.3333333
##
## Group means:
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## setosa 5.006250 3.428125 1.456250 0.250000
## versicolor 6.007692 2.784615 4.376923 1.334615
## virginica 6.740625 2.968750 5.621875 2.015625
##
## Coefficients of linear discriminants:
## LD1 LD2
## Sepal.Length 0.907457 -0.4936054
## Sepal.Width 1.197211 -1.7603466
## Petal.Length -2.187455 1.2776671
## Petal.Width -2.815162 -3.1631095
##
## Proportion of trace:
## LD1 LD2
## 0.9907 0.0093
A predição pode ser realizada através da função genérica stats::predict.
Pode-se fazer um gráfico bivariado para melhorar a apresentação.

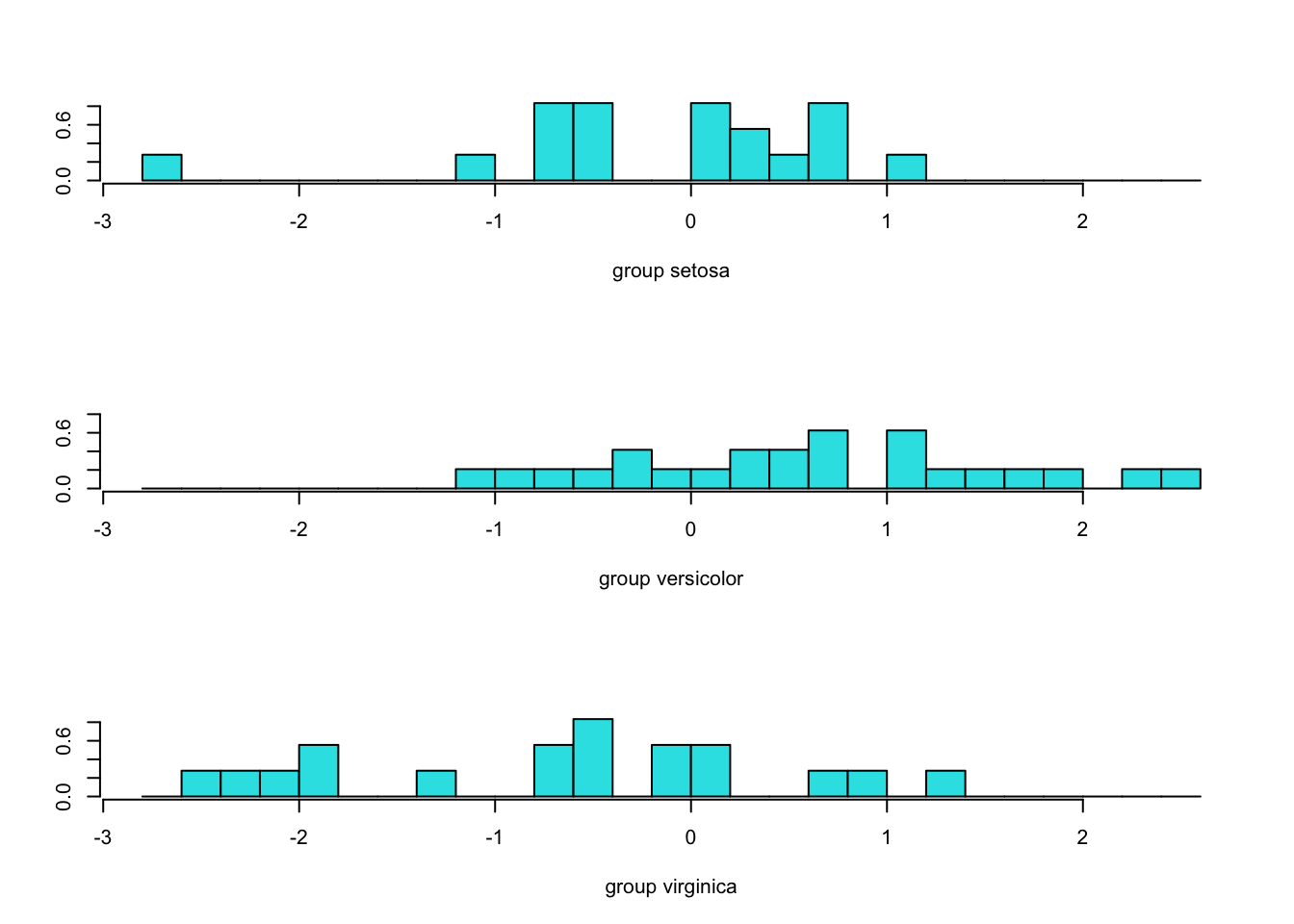
Pode-se criar gráficos por discriminante linear para avaliar a discriminação gerada.

Note que a separação da primeira discriminante linear é muito mais acurada do que a segunda.
Pode-se calcular a acurácia da predição do modelo. Pode-se calcular a proporção de atribuições corretas total e por categoria.
##
## setosa versicolor virginica
## setosa 18 0 0
## versicolor 0 23 1
## virginica 0 0 18## Confusion Matrix and Statistics
##
##
## setosa versicolor virginica
## setosa 18 0 0
## versicolor 0 23 1
## virginica 0 0 18
##
## Overall Statistics
##
## Accuracy : 0.9833
## 95% CI : (0.9106, 0.9996)
## No Information Rate : 0.3833
## P-Value [Acc > NIR] : < 2.2e-16
##
## Kappa : 0.9748
##
## Mcnemar's Test P-Value : NA
##
## Statistics by Class:
##
## Class: setosa Class: versicolor Class: virginica
## Sensitivity 1.0 1.0000 0.9474
## Specificity 1.0 0.9730 1.0000
## Pos Pred Value 1.0 0.9583 1.0000
## Neg Pred Value 1.0 1.0000 0.9762
## Prevalence 0.3 0.3833 0.3167
## Detection Rate 0.3 0.3833 0.3000
## Detection Prevalence 0.3 0.4000 0.3000
## Balanced Accuracy 1.0 0.9865 0.9737Exercício 9.8 Ajuste os modelos LDA, QDA, MDA e FDA no banco de dados wine descrito a seguir.
9.10.1.1 Análise Discriminante Linear com penalidades
O pacote TULIP integra métodos populares baseados em LDA de alta dimensão e fornece ferramentas para classificação linear, semiparamétrica e tensor-variável. As funções estão incluídas para ajuste de modelo, validação cruzada e previsão. Motivados por conjuntos de dados com diversas fontes de preditores estão incluídas funções para ajuste de covariáveis.
9.10.2 Análise Discriminante Quadrática (QDA)
O ajuste do modelo de análise discriminante quadrática pode ser feito pela função MASS::qda.
Exemplo 9.21 Classificando espécies de lírio via QDA.
## Call:
## qda(Species ~ ., data = iris, prior = c(1, 1, 1)/3, subset = itrain)
##
## Prior probabilities of groups:
## setosa versicolor virginica
## 0.3333333 0.3333333 0.3333333
##
## Group means:
## Sepal.Length Sepal.Width Petal.Length Petal.Width
## setosa 5.006250 3.428125 1.456250 0.250000
## versicolor 6.007692 2.784615 4.376923 1.334615
## virginica 6.740625 2.968750 5.621875 2.015625##
## setosa versicolor virginica
## setosa 18 0 0
## versicolor 0 22 0
## virginica 0 2 18## Confusion Matrix and Statistics
##
##
## setosa versicolor virginica
## setosa 18 0 0
## versicolor 0 22 0
## virginica 0 2 18
##
## Overall Statistics
##
## Accuracy : 0.9667
## 95% CI : (0.8847, 0.9959)
## No Information Rate : 0.4
## P-Value [Acc > NIR] : < 2.2e-16
##
## Kappa : 0.9497
##
## Mcnemar's Test P-Value : NA
##
## Statistics by Class:
##
## Class: setosa Class: versicolor Class: virginica
## Sensitivity 1.0 0.9167 1.0000
## Specificity 1.0 1.0000 0.9524
## Pos Pred Value 1.0 1.0000 0.9000
## Neg Pred Value 1.0 0.9474 1.0000
## Prevalence 0.3 0.4000 0.3000
## Detection Rate 0.3 0.3667 0.3000
## Detection Prevalence 0.3 0.3667 0.3333
## Balanced Accuracy 1.0 0.9583 0.9762Para gráficos da QDA veja o link https://stackoverflow.com/questions/63782598/quadratic-discriminant-analysis-qda-plot-in-r.
9.10.3 Análise Discriminante Mista (MDA)
(Robert Tibshirani. Original R port by Friedrich Leisch, Hornik, and code. 2020) propõem uma classe mista de análise discriminante.
Exemplo 9.22 Classificando espécies de lírio via MDA.
## Call:
## mda(formula = Species ~ ., data = train)
##
## Dimension: 4
##
## Percent Between-Group Variance Explained:
## v1 v2 v3 v4
## 95.56 98.61 99.93 100.00
##
## Degrees of Freedom (per dimension): 5
##
## Training Misclassification Error: 0.02222 ( N = 90 )
##
## Deviance: 9.871##
## pred setosa versicolor virginica
## setosa 18 0 0
## versicolor 0 23 1
## virginica 0 1 17## Confusion Matrix and Statistics
##
##
## pred setosa versicolor virginica
## setosa 18 0 0
## versicolor 0 23 1
## virginica 0 1 17
##
## Overall Statistics
##
## Accuracy : 0.9667
## 95% CI : (0.8847, 0.9959)
## No Information Rate : 0.4
## P-Value [Acc > NIR] : < 2.2e-16
##
## Kappa : 0.9495
##
## Mcnemar's Test P-Value : NA
##
## Statistics by Class:
##
## Class: setosa Class: versicolor Class: virginica
## Sensitivity 1.0 0.9583 0.9444
## Specificity 1.0 0.9722 0.9762
## Pos Pred Value 1.0 0.9583 0.9444
## Neg Pred Value 1.0 0.9722 0.9762
## Prevalence 0.3 0.4000 0.3000
## Detection Rate 0.3 0.3833 0.2833
## Detection Prevalence 0.3 0.4000 0.3000
## Balanced Accuracy 1.0 0.9653 0.96039.10.4 Análise Discriminante Flexível (FDA)
(Robert Tibshirani. Original R port by Friedrich Leisch, Hornik, and code. 2020) propõem uma classe flexível de análise discriminante.
Exemplo 9.23 Classificando espécies de lírio via FDA.
## Call:
## fda(formula = Species ~ ., data = train)
##
## Dimension: 2
##
## Percent Between-Group Variance Explained:
## v1 v2
## 99.18 100.00
##
## Degrees of Freedom (per dimension): 5
##
## Training Misclassification Error: 0.02222 ( N = 90 )##
## pred setosa versicolor virginica
## setosa 18 0 0
## versicolor 0 23 0
## virginica 0 1 18## Confusion Matrix and Statistics
##
##
## pred setosa versicolor virginica
## setosa 18 0 0
## versicolor 0 23 0
## virginica 0 1 18
##
## Overall Statistics
##
## Accuracy : 0.9833
## 95% CI : (0.9106, 0.9996)
## No Information Rate : 0.4
## P-Value [Acc > NIR] : < 2.2e-16
##
## Kappa : 0.9748
##
## Mcnemar's Test P-Value : NA
##
## Statistics by Class:
##
## Class: setosa Class: versicolor Class: virginica
## Sensitivity 1.0 0.9583 1.0000
## Specificity 1.0 1.0000 0.9762
## Pos Pred Value 1.0 1.0000 0.9474
## Neg Pred Value 1.0 0.9730 1.0000
## Prevalence 0.3 0.4000 0.3000
## Detection Rate 0.3 0.3833 0.3000
## Detection Prevalence 0.3 0.3833 0.3167
## Balanced Accuracy 1.0 0.9792 0.9881